랜덤포레스트 기법을 이용해 반복수행하는 코드를 만들어 보려고 합니다.
랜덤포레스트 인자중 ntree인자를 바꿔가면서 모델을 만들어보고 가장 좋은 모델은 무엇인지 보려고 합니다.
randomforest함수에 다양한 인자가 있지만 모두 무엇을 뜻하는지 알고싶은데 알기가 힘드네요;;ㅠ
ntree인자와 mtry인자 밖에 모르겠습니다..;;
ntree는 트리의 개수를 몇개 만들지, mtry는 사용하는 feature의 개수를 몇개로 할지 정하는 인자 인것 같습니다.
ntree는 다양하게 바꿔서 사용하는것 같은데 mtry는 모델링에 사용하는 column의 제곱근 개수만큼 보통 지정해 주는것 같습니다.
irisdata를 이용해 ntree의 개수를 바꿔가면서 모델을 만들고 predict값과 real값의 상관계수를 이용해 어떻게 변화하나 보겠습니다.
추가로 rownames가 뒤죽박죽이라 NULL을 넣어 차례대로 넣어줬습니다.
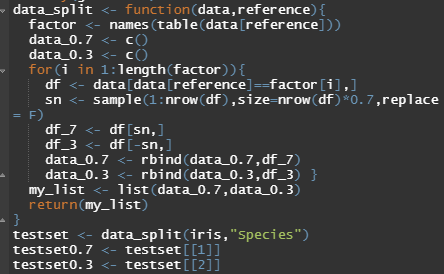
먼저 위 코드를 실행시켜 iris data를 Species기준으로 7:3으로 train과 test set을 나누었습니다.
setosa versicolor virginica 앞과 같은 Species들이 있으므로 각각의 Species가 7:3으로 나뉩니다.
즉, setosa= 7:3 versicolor= 7:3 virginica= 7:3 으로 나누는 거죠
이렇게 나누는 이유는 모두 랜덤하게 나누었다가는 train set에 setosa, versicolor만 들어간다면 virginica를 예측하지 못하기 때문입니다.
여기선 분류를 사용하지 않긴할건데 그래도 그냥 위처럼 나눠봅니다. (알아서 나쁠건 없으니까요)
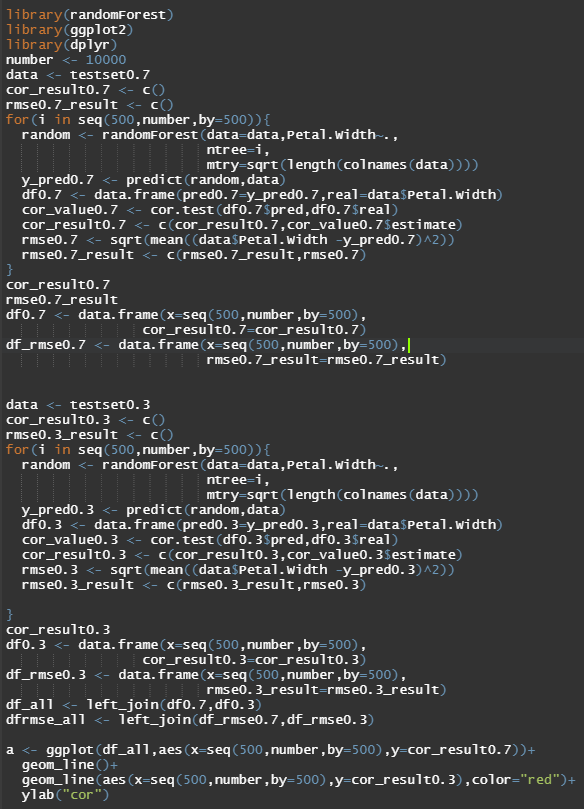
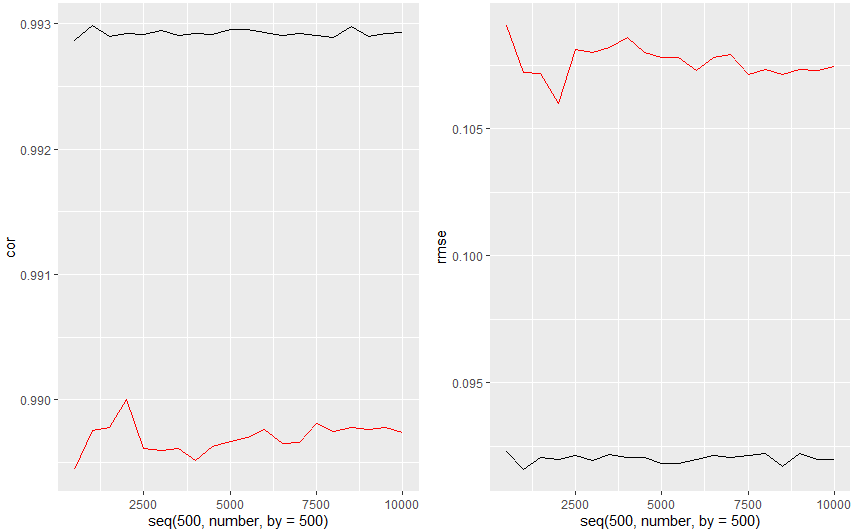
위코드는 randomforest를 ntree인자를 바꿔가며 여러번 반복수행하여 model을 만들고 각 model의 cor,rmse를 구해 ntree가 몇일때 최대의 성능을 내나 확인하기 위해 만들어본 코드 입니다.
rmse식이 저게 맞는지 모르겠네요;;ㅎㅎ cor, rmes가 아닌 다른 지표를 보면서 성능을 측정 할 수도 있겠네요.
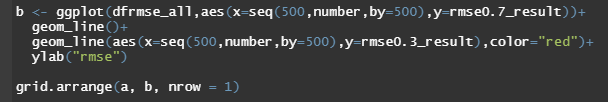
검정은 train set 빨강은 test set data를 이용해 그린겁니다.
cor은 높을수록 rmse는 낮을수록 좋기에 test set의 변화는 그치 않지만 train set의 변화가 ntree=2000에서 가장 좋아보이는 군요
그러므로 ntree=2000에서 모델을 만드는게 가장 model성능이 좋다라고 할 수 있을것 같습니다.
모델을 만들때마다 값이 달라질 수 있는데 이는 set.seed를 사용해 해결해도 될것 같습니다~
install.packages("gridExtra") library(gridExtra) data_split <- function(data,reference){ factor <- names(table(data[reference])) data_0.7 <- c() data_0.3 <- c() for(i in 1:length(factor)){ df <- data[data[reference]==factor[i],] sn <- sample(1:nrow(df),size=nrow(df)*0.7,replace = F) df_7 <- df[sn,] df_3 <- df[-sn,] data_0.7 <- rbind(data_0.7,df_7) data_0.3 <- rbind(data_0.3,df_3) } my_list <- list(data_0.7,data_0.3) return(my_list) } testset <- data_split(iris,"Species") testset0.7 <- testset[[1]] testset0.3 <- testset[[2]] rownames(testset0.7) <- NULL rownames(testset0.3) <- NULL library(randomForest) library(ggplot2) library(dplyr) number <- 10000 data <- testset0.7 cor_result0.7 <- c() rmse0.7_result <- c() for(i in seq(500,number,by=500)){ random <- randomForest(data=data,Petal.Width~., ntree=i, mtry=sqrt(length(colnames(data)))) y_pred0.7 <- predict(random,data) df0.7 <- data.frame(pred0.7=y_pred0.7,real=data$Petal.Width) cor_value0.7 <- cor.test(df0.7$pred,df0.7$real) cor_result0.7 <- c(cor_result0.7,cor_value0.7$estimate) rmse0.7 <- sqrt(mean((data$Petal.Width -y_pred0.7)^2)) rmse0.7_result <- c(rmse0.7_result,rmse0.7) } cor_result0.7 rmse0.7_result df0.7 <- data.frame(x=seq(500,number,by=500), cor_result0.7=cor_result0.7) df_rmse0.7 <- data.frame(x=seq(500,number,by=500), rmse0.7_result=rmse0.7_result) data <- testset0.3 cor_result0.3 <- c() rmse0.3_result <- c() for(i in seq(500,number,by=500)){ random <- randomForest(data=data,Petal.Width~., ntree=i, mtry=sqrt(length(colnames(data)))) y_pred0.3 <- predict(random,data) df0.3 <- data.frame(pred0.3=y_pred0.3,real=data$Petal.Width) cor_value0.3 <- cor.test(df0.3$pred,df0.3$real) cor_result0.3 <- c(cor_result0.3,cor_value0.3$estimate) rmse0.3 <- sqrt(mean((data$Petal.Width -y_pred0.3)^2)) rmse0.3_result <- c(rmse0.3_result,rmse0.3) } cor_result0.3 df0.3 <- data.frame(x=seq(500,number,by=500), cor_result0.3=cor_result0.3) df_rmse0.3 <- data.frame(x=seq(500,number,by=500), rmse0.3_result=rmse0.3_result) df_all <- left_join(df0.7,df0.3) dfrmse_all <- left_join(df_rmse0.7,df_rmse0.3) a <- ggplot(df_all,aes(x=seq(500,number,by=500),y=cor_result0.7))+ geom_line()+ geom_line(aes(x=seq(500,number,by=500),y=cor_result0.3),color="red")+ ylab("cor") b <- ggplot(dfrmse_all,aes(x=seq(500,number,by=500),y=rmse0.7_result))+ geom_line()+ geom_line(aes(x=seq(500,number,by=500),y=rmse0.3_result),color="red")+ ylab("rmse") grid.arrange(a, b, nrow = 1)
'R' 카테고리의 다른 글
| R ) 독학 :: 산점도 알아보기 scatter plot (1) | 2022.05.08 |
|---|---|
| R ) 독학 :: data 시각화 ggplot2 히스토그램 (histogram), 박스 플랏(boxplot) in r -2 플랏 겹치기 플랏 옵션 multi plot, plot option (0) | 2022.05.05 |
| R ) ::독학 dataset 나누기 7:3, 6:2:2 in r (0) | 2022.05.03 |
| R ) 독학 :: data 시각화 ggplot2 히스토그램(histogram), 박스플랏 (box plot) (0) | 2022.04.29 |
| R ) 독학 :: 정규성 검정, 공분산, 상관계수 in r cov, cor, shapiro.test, ad.test (1) | 2022.04.29 |


댓글